Выделения днк из вирусов
Методы выделения ДНК из биологического материала

Выделение ДНК и РНК — важный шаг подготовки проб перед биохимическими и диагностическими процессами. Амплификация, проведение обратной транскрипции, детектирование накопления продуктов амплификации методом ПЦР в реальном времени, клонирование, секвенс, гибридизация, синтез ДНК и т. д., не могут быть выполнены непосредственно на биологических образцах без предварительного выделения (экстракции) нуклеиновых кислот (НК) и их последующей очистки.
Почему так важно получить высококачественную ДНК?
Так как в ветеринарной клинической практике наиболее используемым методом является ПЦР в реальном времени (или, в некоторых случаях, классическая ПЦР), рассмотрим методы выделения НК и их особенности будут рассмотрены именно с точки зрения данного вида анализа.
Механизм ПЦР.
Как известно, ПЦР (полимеразная цепная реакция) представляет собой реакцию синтеза комплементарной цепочки ДНК на ДНК матрице, катализируемую ферментом ДНК-зависимой ДНК-полимеразой. Фермент термостабильный – он может выдерживать высокие температуры, необходимые на этапе денатурации. Оптимум работы полимеразы составляет около 72°C. Для создания оптимальных условий синтеза образец ДНК помещается в так называемую реакционную смесь (РС).
Реакционная смесь состоит:
При должном качестве всех компонентов реакционной смеси реакция может ингибироваться только из-за плохой очистки ДНК. Ингибиторы ПЦР представляют собой вещества, способные повлиять на конформацию фермента и уменьшить его активность, вплоть до полной деактивации. Эти примеси могут оказаться в растворе ДНК по двум причинам:
- Плохая очистка образца от остатков веществ, которые содержались в материале, из которого производилось выделение. Ингибировать ПЦР могут остатки жиров, белков, углеводов, пигментов (для растительных клеток – хлорофиллы, каратиноиды, антоцианы, для животных – гемоглобин и др.). Сюда же относятся и вещества, которые были использованы в качестве транспортной или консервационной среды для образца (ЭДТА, гепарин, формальдегид).
- Плохая очистка образца от реагентов, которые были использованы в процессе выделения. Сильно ингибируют ПЦР детергенты (ПАВ) и денатуранты, такие как SDS (додецилсульфат натрия) и мочевина; спирты и другие неполярные растворители, которые содержатся в экстрагентах и отмывочных буферах (этанол, изопропанол, фенол и др.).
Список некоторых веществ, которые могут негативно повлиять на эффективность ПЦР, показан в таблице 1.
Таблица 1. Некоторые ингибиторы процесса ПЦР
| Ингибитор | Концентрация ингибитора |
| SDS | > 0.005% |
| Фенол | > 0.2% |
| Этанол | > 1% |
| Изопропанол | > 1% |
| Ацетат натрия | > 5 mМ |
| Хлористый натрий | > 25 mМ |
| EDTA | > 0.5 mМ |
| Гемоглобин | > 1 мг/мл |
| Гепарин | > 0.15 i.m/мл |
| Мочевина | > 20 mМ |
| Агароза (при выделении ДНК из геля) | > 1% |
| РНК | > 0,5 мкг/20мкл |
| Реакционная смесь | > 15% |
Из этого можно сделать вывод, что ключевыми факторами успешного выделения НК является грамотная подготовка материала, правильный выбор метода экстракции и точное соблюдение требований протокола выделения.
Какие требования предъявляются к материалу для выделения нуклеиновых кислот?
Прежде всего, биологические образцы должны быть взяты в достаточном количестве для проведения анализа, кроме того, образца должно хватить для проведения повторного анализа. При этом необходимо учитывать вариации содержания НК в различных органах и тканях, а также уменьшение содержания НК из-за деградации, если образец был взят через большой промежуток времени после смерти животного.
Экстракцию нуклеиновых кислот следует начинать как можно скорее после отбора свежих тканей. Ткань может храниться в условиях низкой температуры в холодильнике в течение нескольких дней без риска деградации НК. Однако, если выделение невозможно в ближайшее время, образец должен быть заморожен до температуры от -20°C до -80°C. При этом целесообразно использовать для хранения и перевозки образцов одноразовую пластиковую пробирку или другие плотно закрывающиеся емкости.
После экстракции целесообразно проверить качество выделенной НК, например, измерить концентрацию с помощью флюориметра/спектрофотометра, провести гель-электрофорез или ПЦР со специальными праймерами – факторами элонгации.
Обзор методов выделения НК
Выделение нуклеиновых кислот – один из базовых методов молекулярной биологии. В прошлом процесс выделения и очистки НК был сложным, времязатратным, трудоемким и имел ограничения с точки зрения скорости обработки образцов. На сегодняшний день имеется множество специализированных методик, которые могут использоваться для выделения нуклеиновых кислот с высокой степенью очистки. Их можно условно разделить на две группы: осаждение НК на суспензионный носитель и выделение на колонках. Безусловно, традиционные методы выделения являются надежными и прошли испытание временем, но сейчас на рынке имеется широкий ассортимент товаров, включая полные наборы, содержащие большинство реагентов, необходимых для выделения нуклеиновых кислот. Однако, большинство из них все же требует многократных этапов центрифугирования, которые сопровождаются удалением супернатанта (прим. фаза дисперсионной системы, которая располагается сверху от границы раздела фаз).
В последний годы повысился спрос на автоматические системы. Разработанные для средних и крупных лабораторий, они являются альтернативой трудоемкому ручному методу. Данная технология значительно повышает производительность лаборатории. При этом выход НК и чистота материала, воспроизводимость и прогностичность эксперимента будут максимальными (так же как скорость, точность и надежность анализа в целом), а риск кросс-контаминации (перекрестного заражения) минимизируется.
Фенол-хлороформная экстракция (Guanidinium Thiocyanate-Phenol-Chloroform Extraction).
Обычно протокол выделения нуклеиновых кислот включает следующие этапы: 1.) клеточный лизис, в результате которого разрушаются клеточные структуры и образуется лизат, 2.) инактивацию нуклеаз клетки, таких как ДНКаза и РНКаза, 3.) выделение искомой нуклеиновой кислоты из клеточного дербиса. Экстракция с помощью смеси фенол-хлороформ – один из наиболее старых, но, тем не менее, широко используемых методов выделения нуклеиновых кислот.
Несмотря на то, что фенол – легковоспламеняющееся, коррозионное и токсичное для человека вещество, он способен очень активно денатурировать белки, но не полностью инактивирует РНКазы. Эту проблему можно решить, используя смесь фенол : хлороформ : изоамиловый спирт (в пропорции 25:24:1).
При смешивании фенола и хлороформа образуется двухфазная эмульсия. После центрифугирования гидрофобный слой эмульсии, в котором собираются белки, липиды и углеводы, оседает на дно, а гидрофильный остается сверху. Отбирается верхняя фаза, содержащая ДНК, после чего ДНК осаждают из супернатанта путем добавления этанола или изопропанола в соотношении 2:1 или 1:1 на фоне высокой концентрации солей в лизате. Соли – обычные примеси в образцах нуклеиновых кислот, поэтому их всегда необходимо удалять из образца перед любыми последующими процессами и планируемыми анализами. Следовательно, для обессоливания образца, содержащего нуклеиновую кислоту, требуется одна или несколько стадий отмывки.
Осажденную ДНК выделяют путем повторного центрифугирования, причем избыток солей вымывается с помощью 70%-го этилового спирта, а центрифугирование необходимо для удаления супернатанта – этанола. Затем осажденную ДНК растворяют с помощью ТЕ-буфера или MQ-воды.
FTA-карты.
В данной методике нуклеиновые кислоты выделяются прямо на специальной бумаге, пропитанной смесью реагентов, связывающих ДНК. Для выделения достаточно нанести каплю образца на карту, после чего нанести на нее буфер для выделения и высушить. В дальнейшем, карту можно разрезать на фрагменты и загружать прямо в пробирку для ПЦР. Данный метод подходит только для жидких образцов и не пригоден для ПЦР в реальном времени, однако по времени анализа ему нет равных.
С помощью коммерческих наборов (KITs)
Силикатные носители (Силика, Silica Matrices).
Стеклянные носители (Glass Particle).
Для очистки нуклеиновых кислот используются частицы стекла, стеклянный порошок и стеклянные шарики. Адсорбция нуклеиновых кислот на стеклянный субстрат базируется на тех же принципах, что адсорбционная хроматография.
Очистка нуклеиновых кислот также может осуществляться на силикагеле и стеклянной суспензии в присутствии раствора хаотропных солей.
Диатомит.
Диатомовая земля, известная также как кизельгур или диатомит, содержит до 94% кремния. Применяемая для фильтрации и в хроматографии, она подходит и для очистки плазмидной и ядерной ДНК. Впоследствии ДНК, связанная с диатомитом, вымывается с помощью спиртосодержащего буфера. Потом буфер сливается, а ДНК элюируется.
Очистка нуклеиновых кислот с использованием магнитных микроносителей (Magnetic Bead Based Nucleic Acid Purification).
Анионообменные смолы.
Анионообменные смолы – класс носителей, в которых используется принцип анионного обмена. Он основан на взаимодействии между положительно заряженными группами диэтиламиноэтилцеллюлозы (DEAE) на поверхности смолы и отрицательно заряженными фосфатными группами ДНК-скелета. Анионообменная смола состоит из силикатных гранул с большим размером пор и гидрофильного поверхностного слоя. Большая суммарная площадь поверхности смолы обеспечивает плотное соединение DEAE-групп с НК. Смола работает в широком диапазоне рН (рН=6-9) и/или концентрации солей (0,1-1,6 М). Благодаря этому такие примеси, как белок и РНК вымываются из смолы с при использованием буферов со средней ионной силой, в то время как ДНК остается связанной с ней до этапа элюирования буфером с высокой ионной силой.
Подводя итог, можно заключить, что современный рынок материалов для выделения нуклеиновых кислот характеризуется большим разнообразием. Все методики позволяют получать высококачественные образцы, пригодные для клинических исследований, однако следует учитывать условия работы лаборатории (вид анализов, количество образцов, цены на услуги лаборатории и т.д.), и тогда практический и экономический эффект выбранного метода будет максимальным.

Рассказываем, как устроен первый этап генетического исследования — выделение геномной ДНК из крови, слюны и других биологических образцов.

ДНК — это длинная молекула с отрицательным зарядом: она кодирует инструкцию по сборке отдельных белков, клеток, тканей и целых организмов. ДНК содержится во всех клетках за редкими исключениями: например, зрелые эритроциты млекопитающих теряют ядро с генетическим материалом, чтобы лучше переносить кислород. Поэтому выделить ДНК можно из любой ткани живого или мертвого организма — вопрос только в том, сколько усилий придется затратить.
Сотрудники лаборатории Genotek успешно справлялись с иголками сосны, стволовыми клетками, йогуртами, зародышами лягушек, сердцами мышей и золотистым стафилококком, а автор этой статьи на студенческом практикуме выделяла ДНК из консервированной кукурузы. Но чаще всего источником генетического материала для анализа становится кровь или слюна человека.

Обработка биоматериала и выделение ДНК — это первый этап молекулярно-биологического исследования. Генетический материал хранится в ядрах клеток, которые нужно разрушить, иначе ДНК не выйдет в раствор и будет недоступна для анализа. Из клеток животных часть ДНК можно высвободить простым нагреванием, а вот с клетками растений это не пройдет: они защищены прочной клеточной стенкой из целлюлозы.
Чтобы анализ прошел успешно, важно очистить ДНК от примесей. Примеси ингибируют активность ферментов, которые нужны для подготовки образка к анализу на секвенаторе или чиповом анализаторе.
Самые распространенные ингибиторы в биологических тканях — это гем (компонент эритроцитов, который позволяет им переносить кислород), коллаген в костях и других соединительных тканях, меланин в волосах и коже, ароматические соединения в растительных образцах, миоглобин в тканях мыщц (1, 2). В слюне анализу мешают остатки пищи и консервант, который используется для хранения и перевозки образца: он содержит SDS, ингибирующий работу ферментов. При перевозке SDS защищает ДНК от разрушения, но при попадании в реакционную смесь нарушает работу ДНК-полимеразы.

В 1952 году Херши и Чейз доказали, что наследственная информация содержится в ДНК, а не белках, как считали до этого. Ученые работали с бактериофагом Т2 — вирусом, заражающим кишечную палочку.
Бактериофаг состоит из белковой оболочки и молекулы ДНК внутри нее. Оболочка содержит атомы серы, а ДНК аккумулирует 99% фосфора. Херши и Чейз кормили бактериофагов радиоактивными изотопами фосфора и серы, чтобы меченые атомы включились в состав оболочки и ДНК.
Радиоактивными фагами ученые заражали бактерий и изучали вирусное потомство. Так они выяснили, что дочерние фаги наследуют 30% меченого фосфора и менее одного процента серы.
Результаты эксперимента А. Херши и М. Чейз стали решающим доказательством роли ДНК в передаче наследственной информации.
В лаборатории Genotek выделение ДНК полностью автоматизировано: робот не ошибается и не устает. Процесс выделения происходит в несколько шагов:

При неправильном сборе, некорректном или слишком долгом хранении биологических образцов ДНК в клетках разрушается. Такие образцы подходят для анализа небольших фрагментов генома (например, при установлении отцовства), но непригодны для исследований, где важно прочитать всю генетическую последовательность (секвенирование) или проанализировать одновременно сотни тысяч нуклеотидов в разных участках ДНК (генотипирование на микрочипах).
Важно, чтобы ДНК не разрушилась при перевозке и хранении биообразца. Для этого в пробирку добавляют консервант. Если слюны больше, чем требуется, консервант не сработает. Если слюны мало, ДНК не хватит для анализа. Поэтому все поступившие в лабораторию пробирки оценивают визуально и в случае проблем сразу отправляют на перезабор.
Иногда не получается выделить качественную ДНК из внешне пригодного образца. Тогда лаборатория делает вторую попытку, используя оставшуюся порцию слюны, чтобы исключить технические ошибки. Если она тоже оказывается неудачной, мы приглашаем клиента на перезабор и просит внимательно соблюдать правила сдачи биоматериала: важно, чтобы в слюне оказалось достаточно клеток эпителия щек и не было посторонних примесей.
ДНК, прошедшую контроль качества, направляют на секвенирование или генотипирование на микрочипах.
В наши дни большинство лабораторий используют коммерческие наборы для выделения ДНК, в которых используются спин-колонки, для выделения ДНК и РНК, которые содержат кремнеземную смолу, избирательно связывающую нуклеиновые кислоты, в зависимости от факторов, влияющих на метод экстракции. Наборы для выделения ДНК делают весь процесс намного проще и быстрее, чем старые методы, но недостатком их использования является то, что если вы не понимаете, что находится в черном ящике набора, он делает поиск проблем и ошибок намного сложнее.
Итак, в этой статье я подробно объясню, как работают наборы для извлечения НК и что происходит на каждом этапе, а также о некоторых типичных проблемах, связанных с использованием колонок с силикагелем в экстракции ДНК, которые можно преодолеть или избежать, лишь немного разобравшись.
Первый шаг в экстракции РНК и ДНК: лизис
Формулы лизиса могут варьироваться в зависимости от того, хотите ли вы извлечь ДНК или РНК, но общим знаменателем является лизирующий буфер, содержащий высокую концентрацию хаотропной соли. Хаотропы дестабилизируют водородные связи, силы Ван-дер-Ваальса и гидрофобные взаимодействия. Белки дестабилизируются, в том числе нуклеазы, нарушается связь нуклеиновых кислот с водой, что создает условия для перехода к кремнию. Хаотропные соли включают гуанидин HCl, гуанидинтиоцианат, мочевину и перхлорат лития.
Помимо хаотропов, в буфере для лизиса обычно есть детергенты, которые улучшают солюбилизациею и лизис белка. Также могут быть ферменты, используемые для лизиса, в зависимости от типа образцов. Протеиназа К является одним из них и на самом деле очень хорошо работает в этих денатурирующих буферах; чем больше денатурирован белок, тем лучше работает протеиназа К. Лизоцим не работает при денатурировании, и поэтому обработку лизоцимом обычно проводят перед добавлением денатурирующих солей.
Один комментарий о плазмидных препаратах: лизис сильно отличается от экстракции для выделения РНК или геномной ДНК, потому что плазмида должна быть сначала отделена от геномной ДНК, и если вы добавите хаотропы, вы высвободите все сразу и не будете иметь возможность отделить маленькую кольцевую ДНК от высокомолекулярной хромосомы. Таким образом, в плазмидных препаратах хаотропы не добавляются до тех пор, пока не происходит лизис, и соли не используются для связывания.
После извлечения ДНК происходит очистка: связывание ДНК с колонкой
Хаотропные соли имеют решающее значение для лизиса, а также для связывания нуклеиновых кислот с колонкой. Кроме того, для усиления и влияния на связывание нуклеиновых кислот с диоксидом кремния также добавляют спирт. В большинстве случаев это этанол, реже — изопропанол. Процент этанола и объем имеет большое значение. Слишком много, и вы получите много разложившихся нуклеиновых кислот и мелких частиц, которые будут влиять на показания поглощения света и снижать выход эксперимента. Слишком мало – возникнут при смывании всей соли с мембраны. Важным моментом здесь является то, что этанол влияет на связывание, и добавленное количество оптимизируется для любого набора, который вы используете. Изменение этого шага может помочь изменить то, что вы восстанавливаете, поэтому, если у вас возникли проблемы и вы хотите устранить неполадки восстановления, это может быть шагом для дальнейшей оценки.
Еще один способ диагностики проблем — сохранить поток после связывания и ускорить его, чтобы увидеть, можете ли вы найти искомые нуклеиновые кислоты. Если вы использовали SDS-содержащее моющее средство при лизисе, попробуйте использовать NaCl в качестве осадителя, чтобы избежать загрязнения ДНК или РНК моющим средством.
Отмывка ДНК (или РНК)
Ваш лизат был центрифугирован через силикагелевую мембрану, и теперь ваша экстрагированная ДНК или РНК должна быть связана с колонкой, а примеси, белок и полисахариды должны были пройти. Но мембрана все еще загрязнена остаточными белками и солью. Если образец был взят из растений, на мембране все еще будут присутствовать полисахариды, возможно, некоторые пигменты.
Этапы промывки служат для удаления этих примесей. Обычно есть две промывки, хотя это может варьироваться в зависимости от типа образца. Первая промывка часто содержит небольшое количество хаотропной соли для удаления белка и цветных загрязнений. За этим всегда следует промывка этанолом для удаления солей. Если в препарате недостаточно белка для начала, (плазмидные препараты или очистка продуктов Полимеразная цепная реакция), тогда необходима только промывка этанолом.
Удаление хаотропных солей имеет решающее значение для получения высоких выходов и чистоты нуклеиновых кислот. Некоторые наборы даже дважды промывают колонку этанолом. Если соль останется, элюирование нуклеиновой кислоты будет плохим, и показание A230 (поглощение света с длиной волны 230 нм) будет высоким, что приведет к низким отношениям 260/230. Для ДНК и РНК, не содержащих этанол, вам нужна сухая колонка. После промывки этанолом большинство протоколов имеют стадию центрифугирования для высушивания колонки. Это необходимо для удаления этанола и важно для чистого элюента. Когда для элюирования на мембрану наносят 10 мМ Трис-буфера или воду, нуклеиновые кислоты могут гидратироваться и высвобождаться из мембраны. Если в колонке все еще есть этанол, то нуклеиновые кислоты не могут быть полностью регидратированы.
Пропуск этапа сушки приводит к загрязнению этанолом и низким выходам. Если вы не видете абсорбции этанола на Nanodrop, то он не будет отображаться в ваших показаниях. Основные признаки проблемы состоят в том, что при попытке загрузить образец в агарозный гель ДНК не утонет. Даже при наличии красителя. Другим показателем является то, что если вы поместите образец в -20°C, он не замерзнет.
Экстракция РНК и ДНК, последний рубеж: элюция
Последним этапом в протоколе экстракции ДНК является освобождение чистой ДНК или РНК из силикагеля.
Для приготовления ДНК обычно используют 10 мМ Трис при рН 8-9. ДНК более стабильна при слегка щелочном pH и быстрее растворяется в буфере. И это применимо также для гранул ДНК. Вода имеет тенденцию иметь низкий pH, так как 4-5 и высокомолекулярная ДНК могут не полностью регидратироваться за короткое время, используемое для элюции. Элюция ДНК может быть максимизирована, если позволить буферу сидеть в мембране в течение нескольких минут перед центрифугированием.
РНК элюируется хорошо при слегка кислом pH, и поэтому вода является предпочтительным разбавителем. РНК легко растворяется в воде.
Что еще может пойти не так?
Низкий выход: если выход ДНК/РНК ниже, чем вы ожидали для образца, есть много факторов, которые стоит проанализировать. Обычно это проблема лизиса — неполный лизис является основной причиной низкого выхода. Это также может быть вызвано неправильными условиями связывания. Обязательно используйте свежий высококачественный этанол для разбавления буферов или для добавления на стадии связывания. Возможно, этанол низкого качества или в старые запасы попала вода и они имеют неправильную концентрацию. Если буфер для промывки сделан неправильно, возможно, вы смываете извлеченную ДНК или РНК.
Низкая чистота: если извлеченная ДНК загрязнена белком (низкое соотношение 260/280), возможно, вы начали с слишком большого количества образца, и белок не был полностью удален или растворен. Если ДНК имеет плохое отношение 260/230, проблема обычно заключается в соли из связующего или промывочного буфера. Убедитесь, что этанол высочайшего качества использовался для приготовления промывочных буферов, и, если проблема не исчезнет, дайте колонке дополнительную промывку.
Некоторые образцы имеют гораздо больше ингибиторов по сравнению с другими. Образцы окружающей среды особенно подвержены проблемам с чистотой, потому что гуминовые вещества растворяются во время экстракции. Хьюмик ведет себя подобно ДНК и его трудно удалить из колонки с диоксидом кремния. Для этого типа образца существуют специальные методы для удаления белка и гуминов перед стадией колонки.
Деградация: В основном при экстракции РНК деградация происходит из-за неправильного хранения образца или неэффективного лизиса, при условии что вы элюировали водой без РНКазы. Для экстракции ДНК деградация не является большой проблемой, потому что для ПЦР ДНК можно разрезать, и она работает нормально. Но если вы надеялись не иметь слишком много сдвинутой ДНК, возможно, вы использовали слишком сильный метод лизиса.
Как ученые, конечно, мы хотим точно знать, что происходит с нашими экспериментами, и иметь возможность устранять неполадки, не обращаясь в службу технической поддержки.
Я надеюсь, что эта статья поможет прояснить некоторые научные аспекты метода спин-фильтрации с силикагелем для экстракции РНК и ДНК, чтобы вы могли самостоятельно поставить диагноз и исправить его. Таким образом, когда вы звоните в службу технической поддержки, вы сначала дважды проверили несколько наиболее вероятных причин проблем, и вместо того, чтобы пройти через множество ошибок, вы сможете быстрее найти решение. Даже если это бесплатный набор для замены ДНК!

Впервые нуклеиновые кислоты пытались выделить в середине XIX века, когда ещё практически ничего не было известно об этих молекулах. Однако с момента открытия структуры и свойств ДНК технологии её выделения непрерывно модифицируются и совершенствуются. В данной статье рассматриваются самые распространенные и прогрессивные методики, используемые для экстракции нуклеиновых кислот.
Первое упоминание об использовании этого метода встречается в статье 1967 года 1 , и с тех пор эта технология является одним из самых распространённых способов выделения нуклеиновых кислот.
Суть методики заключается в смешивании клеточного лизата с фенолом, хлороформом и изоамиловым спиртом в пропорции 25:24:1 и последующем перемешивании и центрифугировании смеси. После проведения этих манипуляций получается раствор с двумя фазами: водной и органической, причем все липиды и жиры находятся в органической (нижней) фазе, белки — на границе фаз, а нуклеиновые кислоты — в водной (верхней) фазе 2 (Рис.1). Для повышения чистоты экстракта эти действия повторяют несколько раз. Если раствор будет иметь низкий pH, то ДНК перейдёт в органическую фазу, а РНК останется в водной фазе, что позволяет выделять РНК отдельно от ДНК.
Данный метод используется повсеместно, поскольку он не требует дополнительного сложного оборудования и имеет невысокую стоимость. Однако нуклеиновые кислоты, полученные таким образом, обладают невысоким качеством и зачастую требуют дополнительной очистки. Также эта технология имеет существенно меньший выход нуклеиновых кислот в сравнении с другими методиками.
Помимо качества экстракта, этот метод обладает ещё несколькими недостатками: он требует сложных манипуляций, которые могут привести к контаминации и потере образца, а сам процесс трудно автоматизировать. Также весь протокол занимает достаточно много времени 3 .
Выделение на спин-колонках
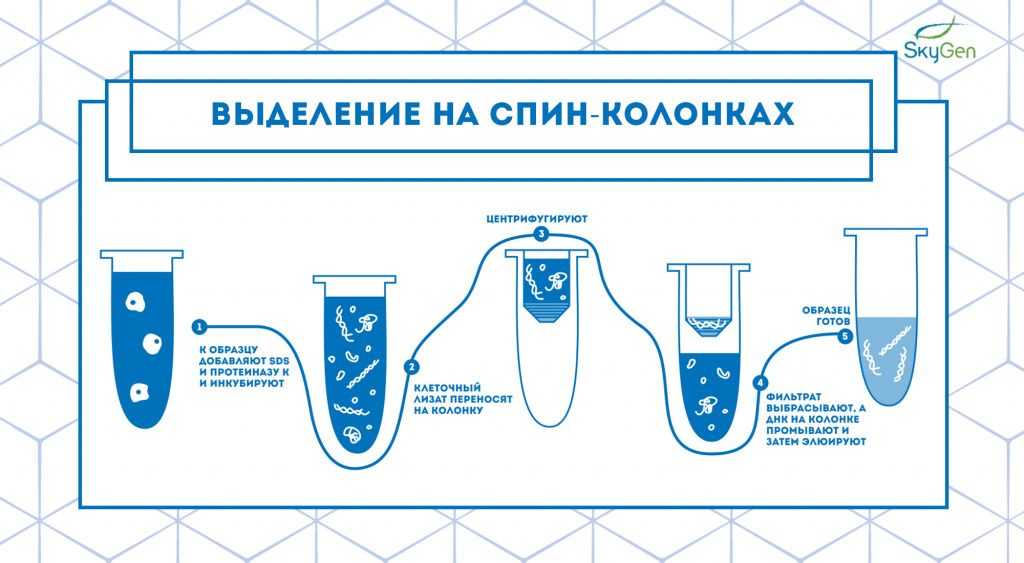
Рис.2. Схема протокола выделения на спин-колонках.
Технология выделения на спин-колонках — это усовершенствованный метод экстракции на частичках силики, предложенный американскими учёными в 1979 году 4 . Они продемонстрировали, что в щелочных условиях и при повышенных концентрациях соли ДНК связывается с силикатами, и это позволяет отделить все остальные компоненты клетки от частиц силики со связанной ДНК. Спин-колонки сконструированы таким образом, что при нанесении клеточного лизата на колонку и последующем центрифугировании ДНК остаётся на колонке, а всё лишнее проходит сквозь неё (Рис.2). Затем ДНК промывают несколько раз и элюируют в пробирку для сбора образца.
Преимущества такого метода заключаются в повышенной чистоте и хорошем качестве выделенных нуклеиновых кислот, высокой воспроизводимости и простоте по сравнению с выделением фенол-хлороформом. Однако также большое количество манипуляций может привести к контаминации, а выделение коротких фрагментов ДНК на спин-колонках может быть затруднено 5 .
Экстракция на спин-колонках может занять от 20 минут в зависимости от биоматериала и сложности его лизиса.
На рынке этот метод представлен большим разнообразием наборов от таких производителей, как Qiagen , Analytik Jena , NEB , ThermoFisher и других. Стоимость одного выделения здесь значительно выше, чем у предыдущего метода, поскольку на каждую реакцию необходима своя колонка, несколько пробирок для сбора фильтрата и элюата и, конечно, реагенты.
Выделение на магнитных частицах
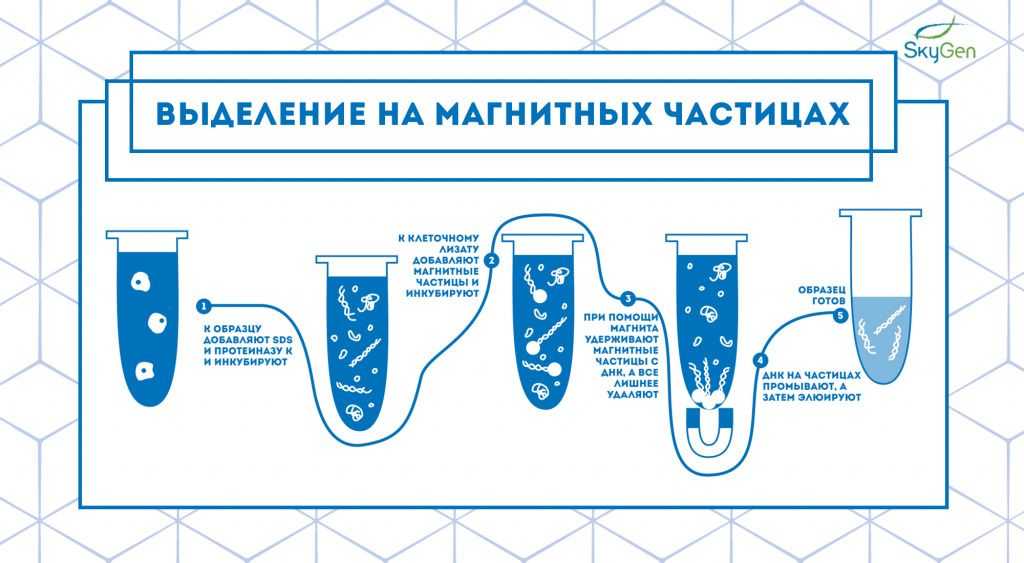
Рис.3. Схема протокола выделения на магнитных частицах.
Спустя 20 лет после появления метода выделения на спин-колонках начинает набирать популярность более быстрый способ выделения на магнитных частицах 6 . Технология этого способа выделения основана на связывании нуклеиновой кислоты с веществом, покрывающим магнитные частицы (целлюлоза, сефадекс, сефакрил, dT-олигонуклеотиды, специфичные олигонуклеотиды и др.). К клеточному лизату добавляют такие магнитные частицы и перемешивают для связывания ДНК с ними. После этого пробирку ставят в магнитный штатив или подносят к магниту, фиксируя таким образом твердую фазу. После отбора супернатанта нуклеиновые кислоты на частицах промывают и элюируют 7 (Рис.3).
Этот метод имеет те же преимущества, что и выделение на спин-колонках, но для экстракции на магнитных частицах не требуется сложное лабораторное оборудование (например, центрифуга). Более того, процесс выделения на магнитных частицах легко автоматизировать, и многие автоматические станции выделения основаны именно на этой методике 3 . Однако здесь также присутствует риск контаминации и потерь образца.
Данный протокол выделения займет немного меньше времени благодаря отсутствию этапов центрифугирования, но количество манипуляций будет примерно таким же. Также стоимость одной реакции обычно выше, чем при выделении на колонках, а панели наборов предоставляют Qiagen , Analytik Jena , ThermoFisher и другие.
Умное выделение (Smart Extraction)
Методики выделения нуклеиновых кислот не перестают совершенствоваться: в 2005 году специалисты из компании Analytik Jena доказали, что для связывания нуклеиновых кислот с неорганической твёрдой фазой можно использовать не только хаотропные соли, но и смесь из хаотропных и нехаотропных солей с низкой ионной силой, эту технологию они назвали Dual Chemistry 8 .
Ферментативное температурно-зависимое выделение
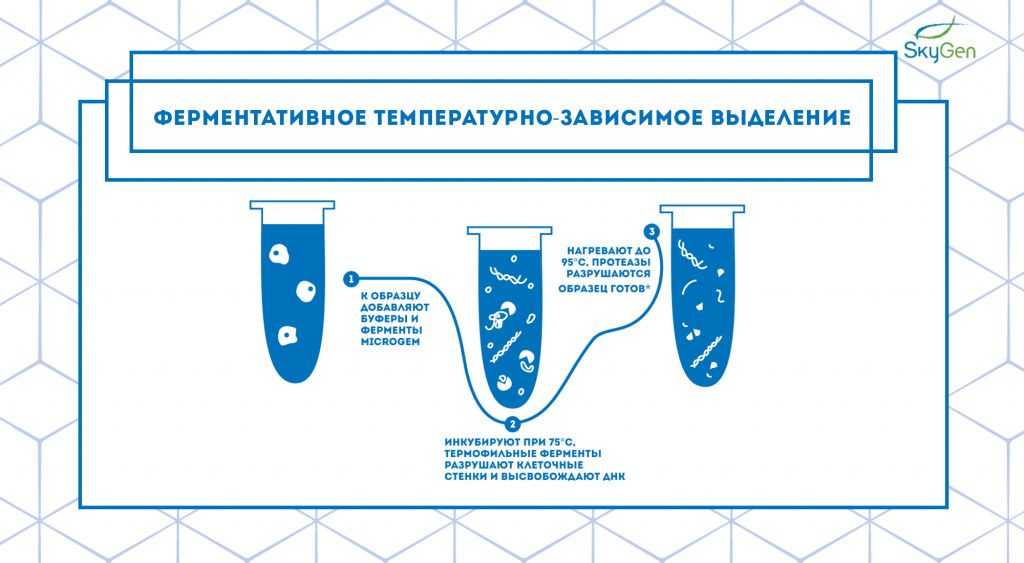
Рис.5. Схема протокола ферментативного температурно-зависимого выделения. Протокол основан на принципе работы наборов для выделения компании MicroGEM. * — несмотря на невысокую степень очистки, образец отлично подходит для дальнейшего использования в ПЦР, секвенировании и STR.
Все вышеперечисленные методики имеют общую лимитирующую стадию — этап лизиса. Во всех технологиях используется SDS и протеиназа K для разрушения клеточных стенок и высвобождения нуклеиновых кислот. SDS является ингибитором ПЦР, именно поэтому необходимы множественные стадии промывки, которые повышают риск контаминации и приводят к потерям образца. Также более сложные для лизиса образцы могут требовать дополнительную долгую и трудозатратную пробоподготовку.
Специалисты из новозеландской компании MicroGEM ликвидировали проблемы, связанные с длительным и сложным лизисом и использованием вредных химикатов, благодаря применению очень эффективной термофильной протеиназы EA1 вместе с мезофильными гидролазами 10 .
Процесс ферментативного температурно-зависимого выделения начинается со смешивания буфера и ферментов с образцом. При последующей инкубации при комнатной температуре гидролазы деградируют клеточные стенки. После этого пробирку нагревают до 75°C, что активирует работу протеиназы EA1, которая разрушает все белки и высвобождает нуклеиновые кислоты. Последующее нагревание до 95°C дезактивирует EA1, и после этого образец готов для дальнейшего исследования (Рис.5). Для особо загрязненных образцов вроде почвы или растений можно добавить этап очистки на колонке для избавления от ингибиторов.
Данная технология оптимальна для работы с малым количеством биоматериала, поскольку нет потерь нуклеиновых кислот. Также эту методику легко автоматизировать, она самая быстрая среди всех упомянутых способов выделения (от 7 минут) и включает меньше всего манипуляций. Стоимость одной реакции невысока, поскольку кроме реагентов не требуется никаких специальных расходных материалов.
Обзор подготовлен при поддержке компании SkyGen — официального дистирибьютора продукции Analytik Jena, MicroGEM, Qiagen, NEB и других производителей.
1. Rae, P. M. M., Barnett, T. R. & Babbitt, D. G. Factors influencing the yield of satellite DNA in extractions from Drosophila virilis and Drosophila melanogaster adults and embryos. BBA Sect. Nucleic Acids Protein Synth. (1976) doi:10.1016/0005-2787(76)90157-X.
2. Patrinos, G. P., Danielson, P. B. & Ansorge, W. J. Molecular Diagnostics: Past, Present, and Future. in Molecular Diagnostics: Third Edition (2016). doi:10.1016/B978-0-12-802971-8.00001-8.
3. Ali, N., Rampazzo, R. D. C. P., Costa, A. Di. T. & Krieger, M. A. Current Nucleic Acid Extraction Methods and Their Implications to Point-of-Care Diagnostics. BioMed Research International (2017) doi:10.1155/2017/9306564.
4. Vogelstein, B. & Gillespie, D. Preparative and analytical purification of DNA from agarose. Proc. Natl. Acad. Sci. U. S. A. (1979) doi:10.1073/pnas.76.2.615.
5. Green, M. R. & Sambrook, J. Molecular Cloning, 3-Volume Set : A Laboratory Manual. Cold Spring Harbour Laboratory Press (2012).
6. Trevor Hawkins. DNA PURIFICATION AND ISOLATION USNG MAGNETIC PARTICLES. United States Pat. (#5,705,628 ) 9, 2989–2997 (1998).
7. Tan, S. C. & Yiap, B. C. DNA, RNA, and protein extraction: The past and the present. Journal of Biomedicine and Biotechnology (2009) doi:10.1155/2009/574398.
Читайте также:


